Introdução
Nesta thread no subreddit de Data Science, um usuário fez o seguinte comentário:
So basically, I was asked to make inference on 10 people and expect those to generalize to the entire study population. I said the study was poorly designed, and that if I made up random numbers we would do a better job of understanding the customer base.
É muito comum pessoas que não são muito familiares com conceitos de inferência estatística ignorar o fato de que tomar conclusões a partir de amostras muito pequenas pode ser bastante perigoso.
Neste artigo escrito por Howard Wainer (Aviso: Link para PDF), são citados dois motivos para que uma equação possa ser perigosa: o fato de elas serem conhecidas e o fato de elas serem desconhecidas. A equação de Moivre, que descreve o desvio padrão da distribuição amostral da média, se encontra no segundo caso:
\(\sigma_{\bar{x}}= \frac{\sigma}{\sqrt{n}}\)
Onde \(\sigma_{\bar{x}}\) é o erro padrão da média, \(\sigma\) o desvio padrão da amostra e \(n\) o tamanho da amostra. Em outras palavras, dado que \(\sigma\) é um parâmetro conhecido, quanto menor o tamanho da amost, maior será erro da estimativa, que corresponde a variação da média amostral em relação à média da população. O paper inteiro é muito bom e fácil de ler, então recomendo a leitura.
O objetivo deste post é um exercício simples para mostrar o efeito do tamanho da amostra na variabilidade dos resultados, mesmo quando se sabem os valores verdadeiros dos parâmetros de toda a população.
Geração dos dados por simulação
library(tidyverse)O primeiro passo é gerar uma população de dados cujos parâmetros são conhecidos. Aqui, duas variáveis aleatórias ´x´ e ´y´ são definidas, em que x é uma variável aleatória e ´y´ deriva de x, com algum ruído:
n <- 1e5
set.seed(123)
x <- rnorm(n, mean = 0, sd = 0.5)
y <- 2 * x + 5 + rnorm(n, mean = 0, sd = 0.25)
df <- data.frame(x = x, y = y)mod_populacao <- lm(y ~ x, data = df)
summary(mod_populacao)##
## Call:
## lm(formula = y ~ x, data = df)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.0972 -0.1685 -0.0002 0.1683 1.0506
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.0013045 0.0007937 6302 <2e-16 ***
## x 1.9986648 0.0015877 1259 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.251 on 99998 degrees of freedom
## Multiple R-squared: 0.9406, Adjusted R-squared: 0.9406
## F-statistic: 1.585e+06 on 1 and 99998 DF, p-value: < 2.2e-16Como esperado, a partir do modelo de regressão obtido acima a partir da população de parâmetros são conhecidos que o processo de geração da variável resposta ´y´ pode ser representado pela equação abaixo:
\(y = 2x + 5 + \epsilon\)
O histograma dos resíduos do modelo é mais uma evidência de que o modelo obtido possui boas propriedades:
hist(resid(mod_populacao),
main = "Distribuição dos resíduos do modelo da população",
xlab = NULL)
Assim, se tirarmos uma amostra da população e criarmos um modelo \(y = f(x)\), os parâmetros do modelo dessa amostra serão os mesmos ou próximos ao da população, correto? Depende de seu tamanho.
Geração das amostras
A função abaixo cria 1000 amostras de tamanho sample_size do dataframe df dos dados da população e, para cada uma das amostras, ajusta um modelo de regressão linear, extrai o coeficiente de x da regressão e retorna um dataframe com os resultados:
coef_x_sample <- function(sample_size){
repl <- purrr::rerun(1000, sample_n(df, sample_size))
vec_coef_x <- repl %>%
map(~ lm(y ~ x, data = .)) %>%
map_dbl(~ coef(.)["x"])
data.frame(size = sample_size, coef_x = vec_coef_x)
}
# exemplo
set.seed(123)
head(coef_x_sample(10))## size coef_x
## 1 10 2.065196
## 2 10 2.320681
## 3 10 2.142100
## 4 10 2.069578
## 5 10 2.036510
## 6 10 1.704862Nas 6 primeiras amostras de tamanho 10, o coeficiente de x foi desde 1,70 a 2,32, mostrando uma certa variabilidade.
O código abaixo repete o processo acima para valores diferentes do tamanho da amostra:
vec_samp_size <- c(5, 10, 15, 20, 50, 75, 100, 150, 200, 250, 500, 750, 1000, 5000)
df_result <- vec_samp_size %>% map_dfr(coef_x_sample)
df_result <- df_result %>%
mutate(size = factor(size, levels = vec_samp_size))Análise dos resultados
Vários gráficos podem ser feitos para ilustrar os resultados da variabilidade do parâmetro da regressão em funcão do tamanho da amostra, como o boxplot abaixo:
df_result %>%
ggplot(aes(x = size, y = coef_x)) +
geom_boxplot() +
scale_y_continuous(breaks = seq(-4, 4, by = 1)) +
labs(x = NULL, y = NULL,
title = "Variabilidade do coeficiente de x em função do tamanho da amostra") +
theme_minimal()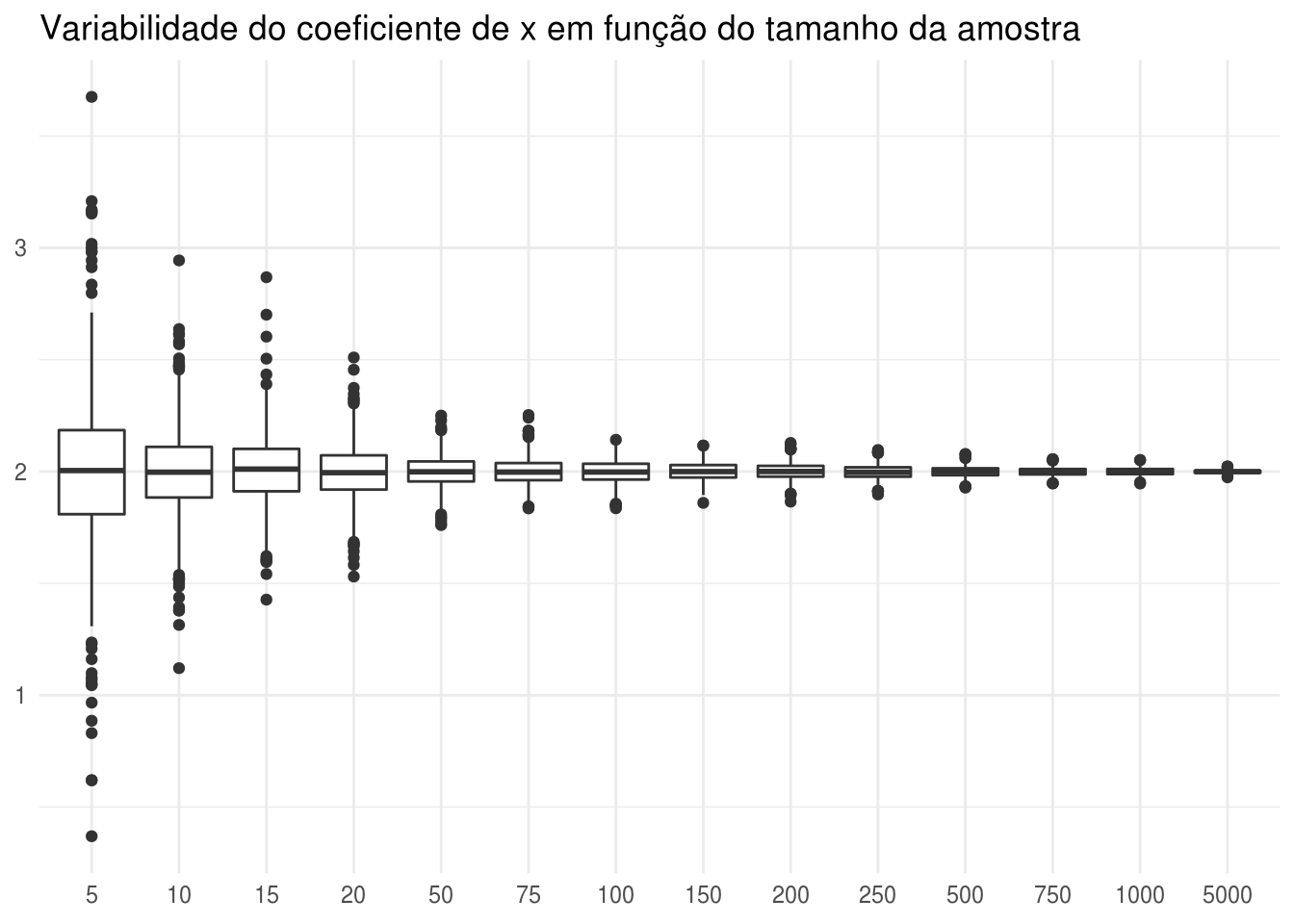
O gráfico acima mostra que, apesar de a mediana ser aproximadamente 2 em todos os tamanhos amostrais testados, a variabilidade é muito maior nas menores amostras. A medida que se aumenta o número de indivíduos na amostra, a distribuição dos resultados converge para o valor verdadeiro. Para amostras de 5 indivíduos, foram observados resultados no seguinte intervalo:
# intervalo para menor tamanho amostral
range(df_result$coef_x[df_result$size == "5"])## [1] 0.3693973 3.6749739# intervalo para maior tamanho amostral
range(df_result$coef_x[df_result$size == "5000"])## [1] 1.973962 2.022880Ou seja, mesmo que uma amostra seja oriunda de uma população descrita por parâmetros já determinados, usar resultados de uma amostra pequena pode levar a conclusões erradas.